Молекулярная филогенетика | это... Что такое Молекулярная филогенетика? (original) (raw)

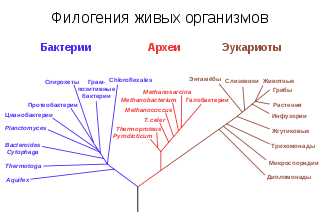
Филогенетическое дерево, построенное с помощью методов молекулярной филогенетики
Молекулярная филогенетика — способ установления родственных связей между живыми организмами на основании изучения структуры полимерных макромолекул — ДНК, РНК и белков. Результатом молекулярно-филогенетического анализа является построение филогенетического дерева живых организмов.
Близкое родство между живыми организмами обычно сопровождается большой степенью сходства в строении тех или иных макромолекул, а молекулы не родственных организмов сильно различаются между собой. Молекулярная филогения использует такие данные для построения филогенетического древа, которое отражает гипотетический ход эволюции исследуемых организмов. Возможность анализировать и подробно изучать эти молекулы появилась только в последние десятилетия XX века.
Молекулярная филогенетика оказала сильнейшее влияние на научную классификацию живых организмов. Методы работы с макромолекулами стали доступны биологам самых различных специальностей, что привело к лавинообразному накоплению новой информации о живых организмах. На основании этих данных старые предположения об эволюции живых организмов пересматриваются. Описывают новые группы, в том числе, выделяемые только на основе молекулярно-филогенетических данных.
Содержание
Методы построения филогенетических деревьев в молекулярной филогенетике
Существует большое количество методов построения филогении на основании молекулярных данных. Их можно подразделить на два типа:
- Методы использующие оценку генетических дистанций
- Методы использующие анализ дискретных признаков
Методы основанные на анализе генетических дистанций
Данная группа методов базируется на данных о генетических дистанциях. Общий принцип заключается в попарном сравнении объектов и построении матрицы дистанций, которая затем используется для построения филогенетического дерева.
UPGMA
Метод попарного внутригруппового невзвешенного среднего (Unweighted Pair Group Method with Arithmetic Mean, UPGMA) считается одним из самых простых. В нынешнем виде метод был представлен в работе Sneath и Sokal 1973 года. Первоначально использование в филогенетике связано с построением фенограмм по морфологическим признакам. Необходимым условием использования метода является постоянная скорость эволюции исследуемых нуклеотидных последовательностей. При неравномерной скорости эволюции последовательностей (несоответствие модели молекулярных часов) метод UPGMA может приводит к ошибкам в топологии дерева.
Алгоритм
На первом этапе в матрице дистанций находят два таксона с наименьшим значением дистанции. Эти два таксона объединяются в один кластер (или составной таксон). Поскольку в рамках данного метода принимается равномерность скорости молекулярной эволюции, то точка ветвления (дивергенции) находится на половине от генетической дистанции между двумя этими таксонами. В дальнейшем этот кластер из двух таксонов считается единым целым. Матрица дистанций пересчитывается, при этом принимается, что расстояние между составным таксоном и остальными таксонами равно:
duk = (du1k+du2k)/2
где d — генетическая дистанция, u — композитная последовательность, u1 и u2 — элементы композитной последовательности, k — таксоны не входящие в композитную последовательность
Затем снова выбираются два таксона имеющие наименьшую генетическую дистанцию, объединяются в кластер и строится новая матрица дистанций и так далее.
Neighbor-joining
Minimum evolution
Метод базируется на предположении, что наиболее вероятным будет дерево с наименьшем количеством эволюционных событий. Принципом данного метода является вычисление длин ветвей (которая отражает количество эволюционных событий) всех возможных топологий деревьев:
 , где bi — оценка длин i-той ветви, T — общее количество ветвей
, где bi — оценка длин i-той ветви, T — общее количество ветвей
В качестве наилучшего, выбирается дерево с наименьшей длиной ветвей. Если для нескольких деревьев с разной топологией длины ветвей не имеют статистически значимых различий, то эти деревья рассматриваются как равновероятные.
Методы основанные на анализе дискретных признаков
Maximum parsimony
Maximum likelihood
Bayesian inference
Литература
- Лукашов В.В. Молекулярная эволюция и филогенетический анализ. — М.: БИНОМ, 2009. — ISBN 978-5-9963-0114-0
- Ней М., Кумар С. Молекулярная эволюция и филогенетика. — Киев: КВЩ, 2004. — ISBN 966-7192-53-9